
| Cohort | N | 측정 플랫폼 | 시료 보관 형태 |
|---|---|---|---|
| GSE13861 | 64 | 마이크로어레이 (Illumina) | 동결신선조직 |
| GSE84437 | 433 | ||
| GSE15459 | 200 | 마이크로어레이 (Affymetrix) | |
| GSE62254 | 300 | ||
| TCGA | 262 | 시퀀싱 (Illumina) |
위암의 mRNA 유전자 발현 빅데이터를 사용해 위암에서 특이적인 유전자들의 발현 특징을 찾기 위해서 인공지능을 활용하여 분석했습니다.
특히 해당 연구개발에서 사용된 분석기법에는 인공지능의 범주인 머신러닝, Representation 러닝이 사용됐습니다.
이는 통계적인 구조와 수학적 프레임워크로 2만개 가량 유전자 데이터의 발현 특징(표현; feature)을 학습해 자동 분류하는 규칙으로 만드는 것입니다.
본 연구에서 사용된 Representation 러닝과 딥러닝 방법으로 음수미포함행렬분해 방법(NMF; non-negative matrix factorization),
유전자상관관계네트워크 분석(WGCNA; weighted gene co-expression network analysis), 유전자 셋 분석(GSA; gene-set enrichment analysis), GOEA(gene oncology enrichment analysis),
계층적 클러스터링(Hierarchical Clustering) 등이 있습니다. 이를 통해 발굴한 위암 대표적 유전자의 발현 특징과 예후 및 항암제 생존이익 사이의 연관 분석을 시행하여 위암의 예후 예측 및
항암제 적합성을 제공하는 기술을 개발했습니다.


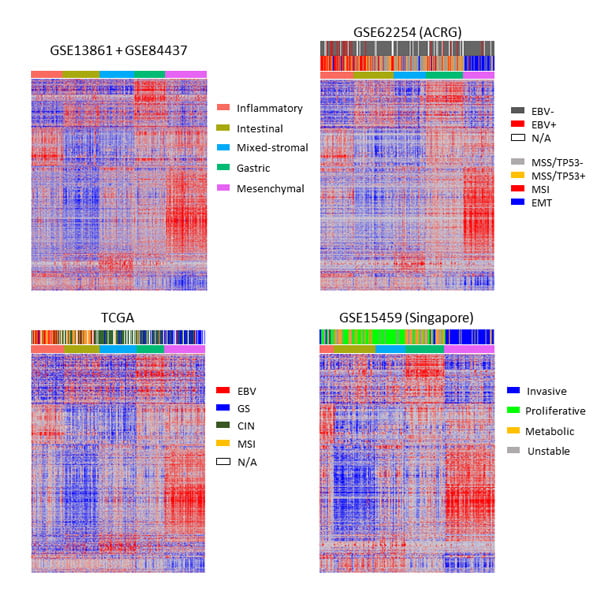
위암 수술 환자의 신선동결조직을 마이크로어레이(microarray) 플랫폼으로 측정한 결과 데이터를 가지고 NMF Clustering 과정을 실행해,
5개의 위암 분자아형(GC molecular subtype)으로 분류하고 GSEA(gene set enrichment analysis)로 아형별 특징적인 유전자들에 따라 annotation했습니다.
분류된 5개의 subtype은 i) 면역적 특징을 갖는 군인 Inflammatory subtype, ii) 장상피분화와 세포증식 관련 유전자 고발현군인 Intestinal subtype, iii) 위
점막 특이적 유전자 고발현군인 Gastric subtype, iv) 이성질적(heterogeneous) 전이-증폭(transit-amplifying) 특징을 갖는 군인 Mixed-stromal subtype, v)
EMT와 mesenchymal 특징을 갖는 군인 Mesenchymal subtype입니다. 이는 다른 3개의 독립적인 공공 코호트(GSE15459, TCGA, GSE62254)에서도 유효함을
확인했습니다.
※ NMF : 음수 미포함 행렬 분해(Non-negative matrix factorization, NMF) : 유전자 발현 데이터를 두 행렬의 곱으로 분해하는 수학적 알고리즘. 군집 성질이 있어
유전자 발현 데이터를 그룹화하고, 군집된 데이터의 대표적인 유전자를 찾는 데에 응용할 수 있다.
Brunet JP, Tamayo P, Golub TR, Mesirov JP. Metagenes and molecular pattern discovery using matrix factorization.
Proc Natl Acad Sci U S A. 2004 Mar 23;101(12):4164-9.
Lei Z, et al., Identification of molecular subtypes of gastric cancer with different responses to PI3-kinase inhibitors and 5-fluorouracil.
Gastroenterology. 2013 Sep;145(3):554-65. Cancer Genome Atlas Research Network. Comprehensive molecular characterization
of gastric adenocarcinoma. Nature. 2014 Sep 11;513(7517):202-9.
Cristescu R, et al., Molecular analysis of gastric cancer identifies subtypes associated with distinct clinical outcomes.
Nat Med. 2015 May;21(5):449-56.


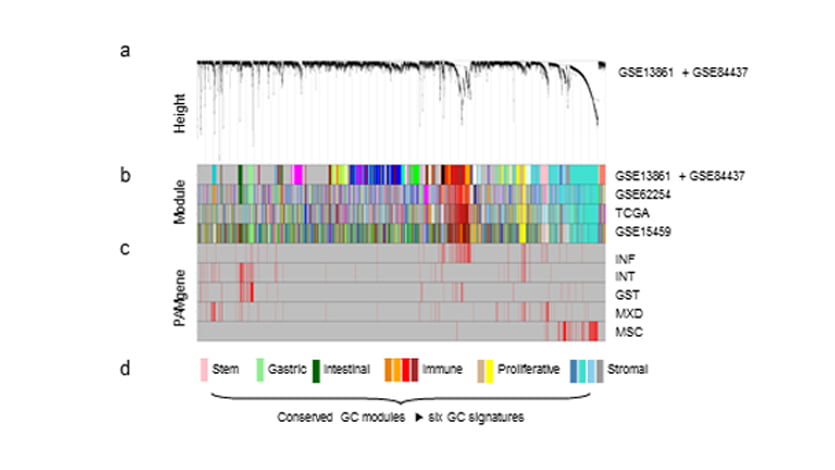
WGCNA 분석을 통해 유전자들 간에 clustering을 했습니다. 이를 바탕으로 위암의 유전체 레벨을 구조화한 34개의 gene-module(module; clusters
of highly interconnected genes; M01부터 M34)을 발굴했습니다. 34개의 gene-module과 5개의 subtype을 비교해 상관관계 (|ρ| > 0.3, ρ=상관계수)를 분석했으며,
5개의 subtype과 연관성이 높은 21개 module들을 선별했습니다.
※ WGCNA(Weighted Gene Co-expression Network Analysis): 유전자 발현 간 상관관계(correlation)를 기반으로 유전자들 간의 네트워크를 파악해 주는 분석방법
Langfelder P, Horvath S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 2008 Dec 29;9:559.


이후에 이들 21개 module간의 상관관계(Inter-modular correlation)를 분석하여 위암의 주요 특성을 가진 6개의 GC molecular feature에 대하여 그룹핑했습니다.
Spearman 상관계수로 위암의 21개 주요 gene-module과 5개 subtype 사이의 상관관계를 분석하였는데, 상관관계가 높을수록 빨간색에 가까우며 상관관계가 낮을수록
파란색에 가깝게 나타납니다.
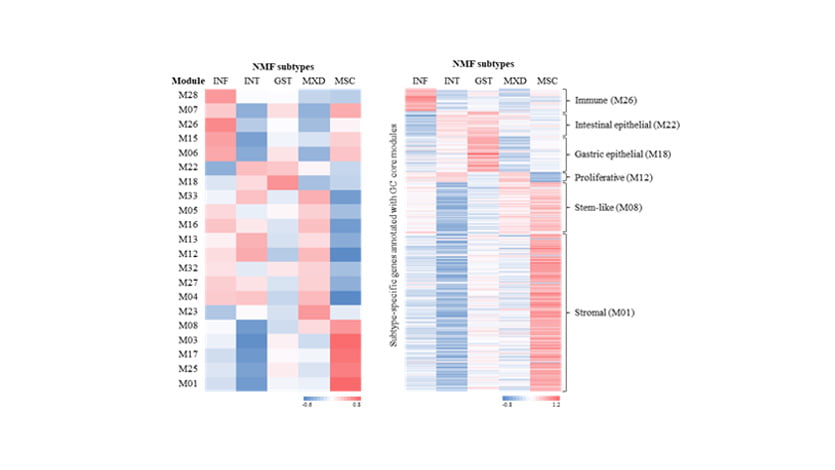
① Immune (M06, M07, M15, M26, M28) / ② intestinal epithelial (M22) / ③ gastric epithelial (M18)
④ proliferative (M04, M05, M12, M13, M16, M23, M27, M32, M33) / ⑤ stem-like (M08) / ⑥ stromal (M01, M03, M17, M25)


최종 예후 관련 표적 유전자 선정과 구분 알고리즘 개발을 위해서 앞선 위암 subtyping 구분에 대한 기존 연구 결과를 근거로 Supervised 방법을 사용했습니다.
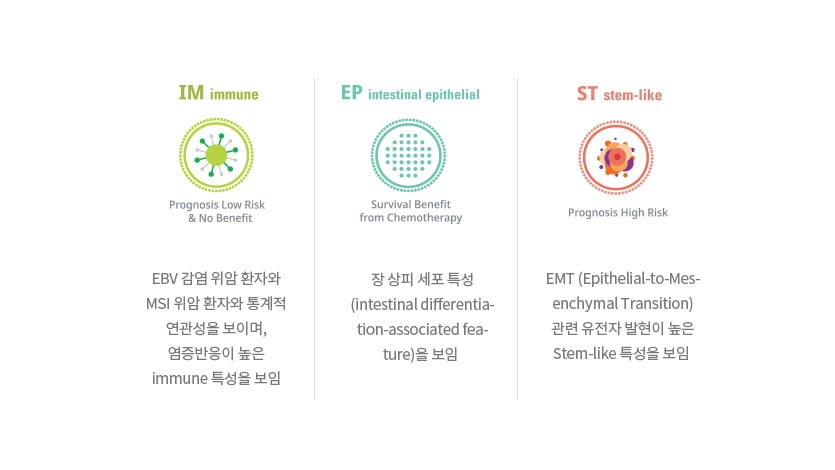
이러한 연구 결과를 바탕으로 위암을 분류하는 주요 선별자로 3가지 핵심 생물학적 특징(Immune, Stem-Like, Epithelial feature)을 선정했습니다.
3가지 핵심 feature의 특징은 다음과 같습니다.


Sampling method와 측정 Platform간 호환성 검정을 통한 유전자 선별: IM, ST, EP feature를 대표하는 M26(Immune module: 48개 유전자),
M08(Stem-like module: 131개 유전자), M22(Intestinal epithelial module: 60개 유전자)가 선택되었습니다.
이들 유전자 중 최종 예측 테스트 후보 유전자를 선별하기 위해서 본 연구에서는 환자의 FFPE 잔여 검체와 qPCR 기반 측정 플랫폼 사용을 개발 목표로 하고 있기 때문에,
발굴된 후보 유전자들에 대하여 sample preparation 및 측정 platform 간의 호환성 분석이 필요했습니다. 따라서 검체는 신선동결조직과 파라핀포매 샘플 사이와,
측정 플랫폼은 마이크로어레이와 qPCR 방법 사이의 호환성을 분석했습니다. 위암 환자의 샘플을 통해 위의 샘플링 방법과 측정 방법에 적용시켜 얻어진
유전자 발현 패턴으로 상관관계를 분석해 기준(Pearson의 상관계수 ρ >0.5)에 안정적으로 부합하는 후보 유전자를 선정했습니다.(12개의 후보 Classifier gene)
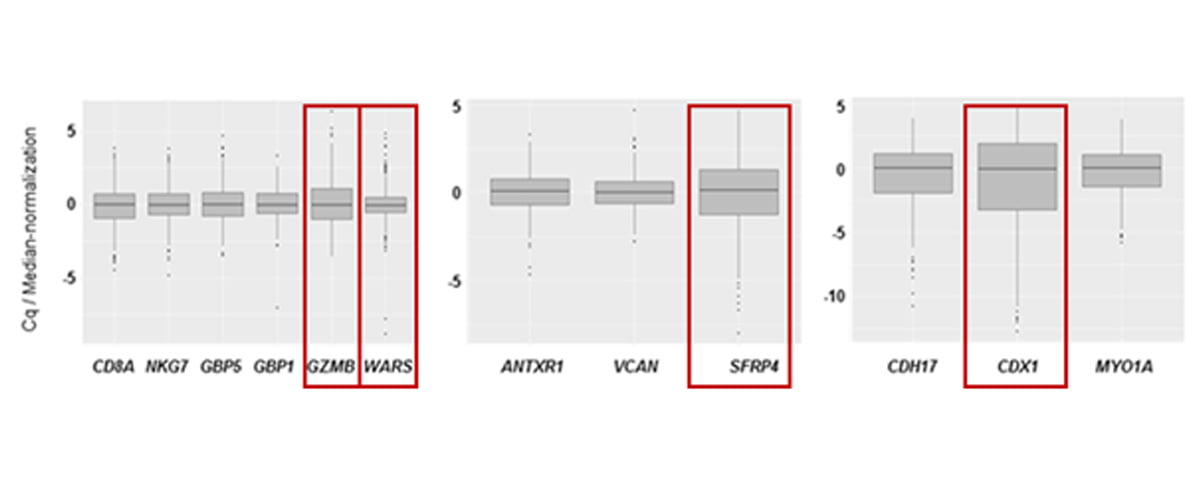
마지막으로 Dynamic range(유전자 발현량 범위) 폭이 넓은 유전자 3개(GZMB, SFRP4, CDX1)와 Immune high 샘플의 높은 변별력을 위하여 면역 관련 Classifier 중
하나인 감염 관련 유전자 1개(WARS)를 추가 선정하여 최종 유전자 Classifier를 4개로 정의했습니다. 최종 선정된 4개의 Classifier gene(GZMB, SFRP4, CDX1, WARS)을
선정했습니다.


유전자 발현량을 보정하기 위한 참고유전자 선정을 위해서 위암에 구체적으로 기 적용되는 참고유전자 (reference gene)에 대해서 논문을 통해 문헌조사를 실시했습니다.
또한 현재 상용화된 고형암 관련 제품에서 활용되고 있는 참고유전자를 조사했습니다.
이들을 근거로 선정된 유전자가 임상샘플의 참고유전자로서 적격한지의 여부를 선행연구에서 검정하여 최종 선정했습니다.
위의 근거를 바탕으로 일차적으로 총 8개의 참고유전자를 후보(ACTB, ATP5E, HPRT1, PGK1, GPX1, RPL29, UBB, and VDAC2)로 선정했고,
FFPE 일상적 잔여 검체(30개)에서 조합하였을 때 가장 변이도가 작은 5개의 참고유전자를 최종 선정했습니다(ACTB / ATP5E / GPX1 / UBB / HPRT1).